Depuis l’informatisation des services hospitaliers, de nombreuses données sont disponibles pour la recherche dans le milieu de la santé.
Ces données sont stockées dans des entrepôts de données de santé (EDS), au sein des hôpitaux.
A l’heure actuelle, pour pouvoir utiliser ces données pour la recherche, il faut avoir des connaissances poussées en programmation. En effet, manipuler ces données nécessite des compétences en SQL, R et Python notamment.
La réalisation d’études sur EDS nécessite une collaboration étroite entre cliniciens, data scientists et statisticiens.
Il existe des plateformes de travail collaboratif telles que Jupyter Notebook1, où le code est mêlé à l’affichage des résultats de l’exécution de ce code. Néanmoins, cette plateforme nécessite des connaissances en programmation et a des fonctionnalités limitées, n’étant pas créée dans le but précis de la création d’études sur données d’EDS.
D’autre part, il existe des logiciels d’analyse statistique, avec des interfaces graphiques dites “no-code”, permettant de réaliser des analyses statistiques sans nécessiter de connaissances a priori en programmation. Ce type de logiciel ne permet habituellement pas de modifier le code, ce qui est limitant pour le data scientist et le développement de nouvelles fonctionnalités.
C’est pourquoi LinkR a été créée.
C’est une application web qui permet :
Ainsi, elle permet aux différents intervenants impliqués dans la recherche médicale sur EDS de travailler de façon collaborative, sur une plateforme commune.
Du fait de cette plateforme commune adaptée aux cliniciens et aux data scientists, les études peuvent être réalisées beaucoup plus vite, et avec l’apport de la spécificité de chacun : l’expertise médicale du clinicien et l’expertise des données du data scientist.
LinkR permet l’analyse de données provenant de différentes sources :
Une fois ces données importées dans LinkR, vous pouvez créer des études. Chaque étude comprendra un ou plusieurs subsets de patients.
Organisation des données au sein de LinkR
Une fois une étude créée, il faut la configurer.
Le principe est simple : une étude comporte une page “Données individuelles” et une page “Données agrégées”.
Dans la page des données individuelles, je créé, selon mes besoins, un template de dossier médical.
Par exemple, si mon étude porte sur les pneumopathies bactériennes, je vais créer :
Avantages :
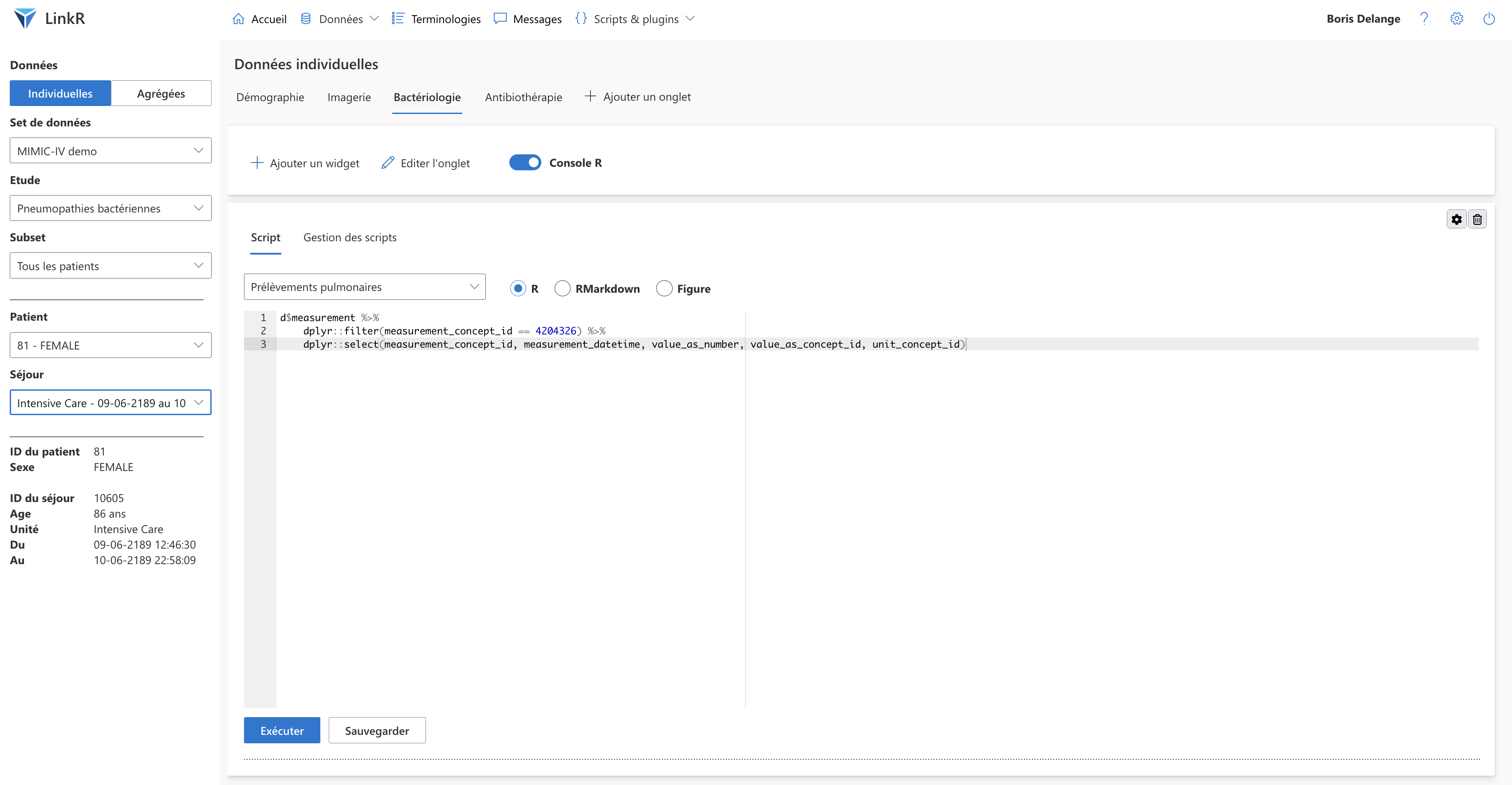 Onglets de données individuelles avec un plugin "Console R"
Onglets de données individuelles avec un plugin "Console R"
Une fois que cela est fait, nous pouvons poursuivre notre étude via l’analyse des données agrégées.
De la même façon, je créé des onglets, correspondant aux différentes étapes de mon étude :
Chacun de ces exemples correspond à ce qu’on appelle un plugin : c’est un script permettant d’ajouter une fonctionnalité à l’application.
Nous aurons donc un plugin “Flowchart”, un plugin “Bibliographie” etc.
Avantages :
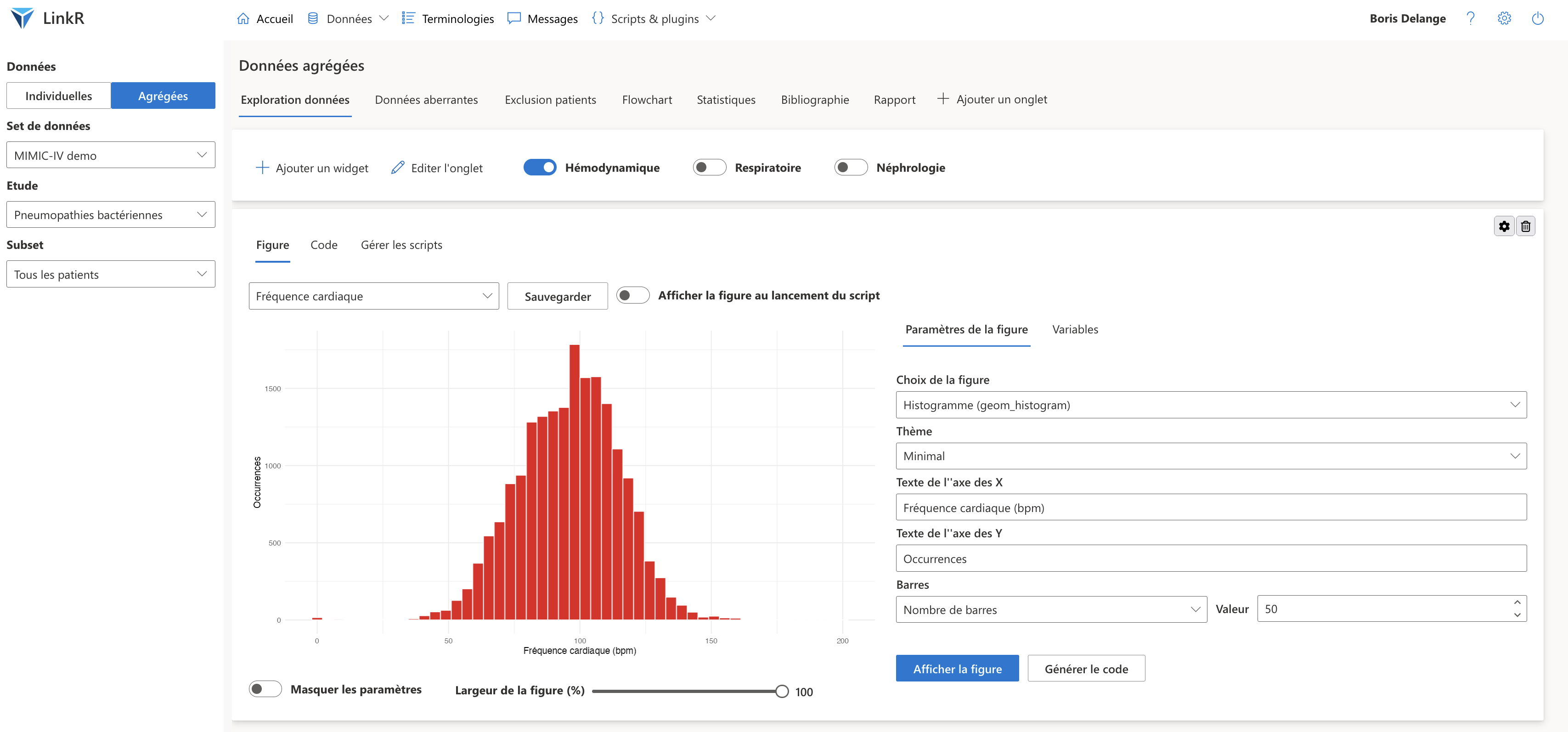 Onglets de données agrégées avec un plugin "Figure (ggplot2)"
Onglets de données agrégées avec un plugin "Figure (ggplot2)"
Les différents intervenants peuvent communiquer via l’onglet “Messages”.
Ainsi :
Le code peut être exécuté au sein des messages.
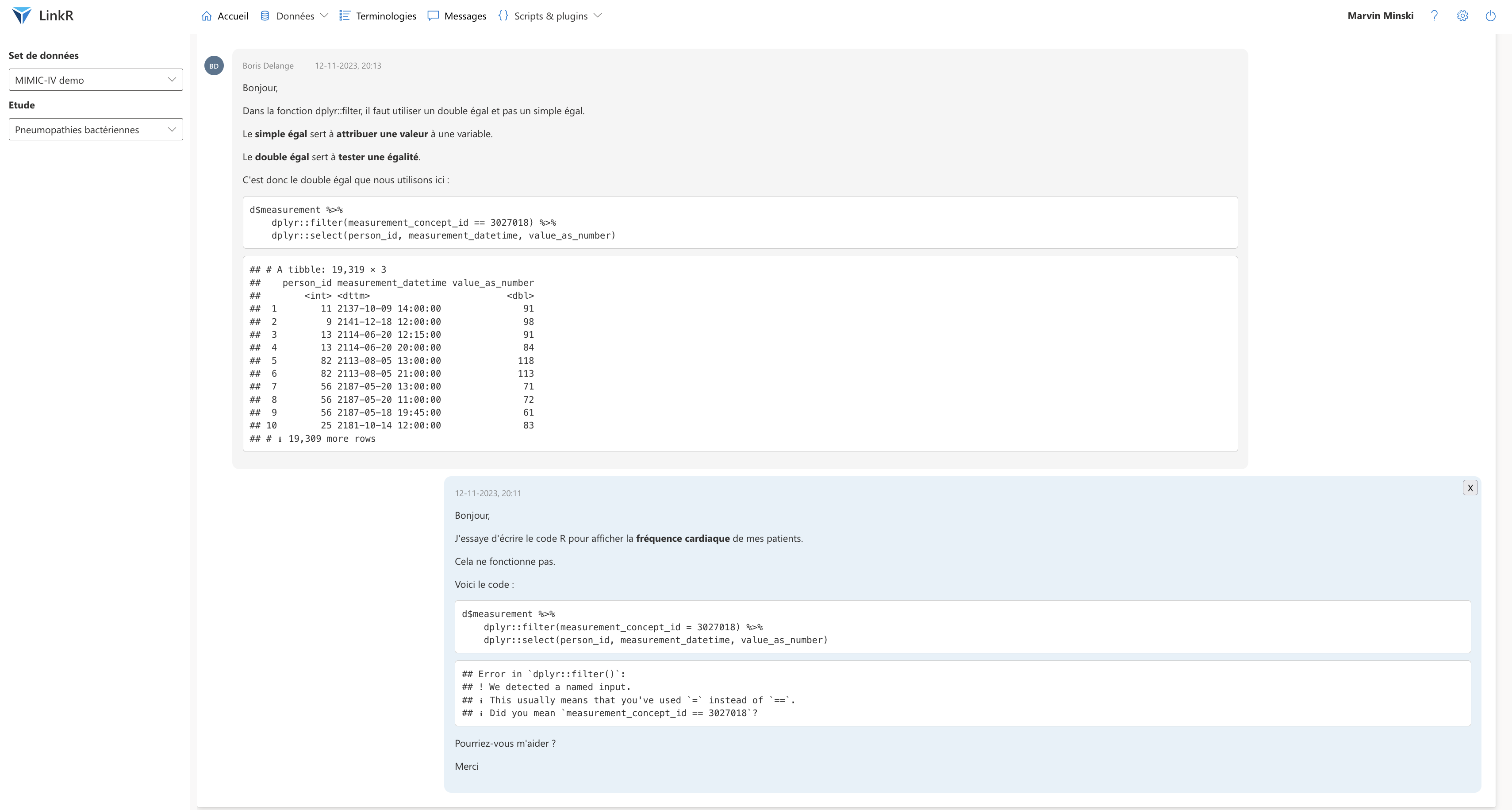 Echange de messages entre les membres de l'étude "Pneumopathies bactériennes"
Echange de messages entre les membres de l'étude "Pneumopathies bactériennes"
LinkR s’appuie sur le modèle de données commun OMOP, qui est un modèle utilisé par de nombreux hôpitaux dans le monde.
Ceci permet l’interopérabilité des données, il est ainsi possible de réaliser des études multicentriques facilement, en ne codant l’étude qu’une fois.
LinkR intègre des outils d’alignement des concepts, permettant de faire correspondre les concepts contenus dans différentes bases de données.
Par exemple pour faire correspondre le concept “123456 - Fréquence cardiaque” du centre 1 au concept “456789 - FC (bpm)” du centre 2.
Il est également possible de partager tout ce que vous développez au sein de LinkR.
A savoir :
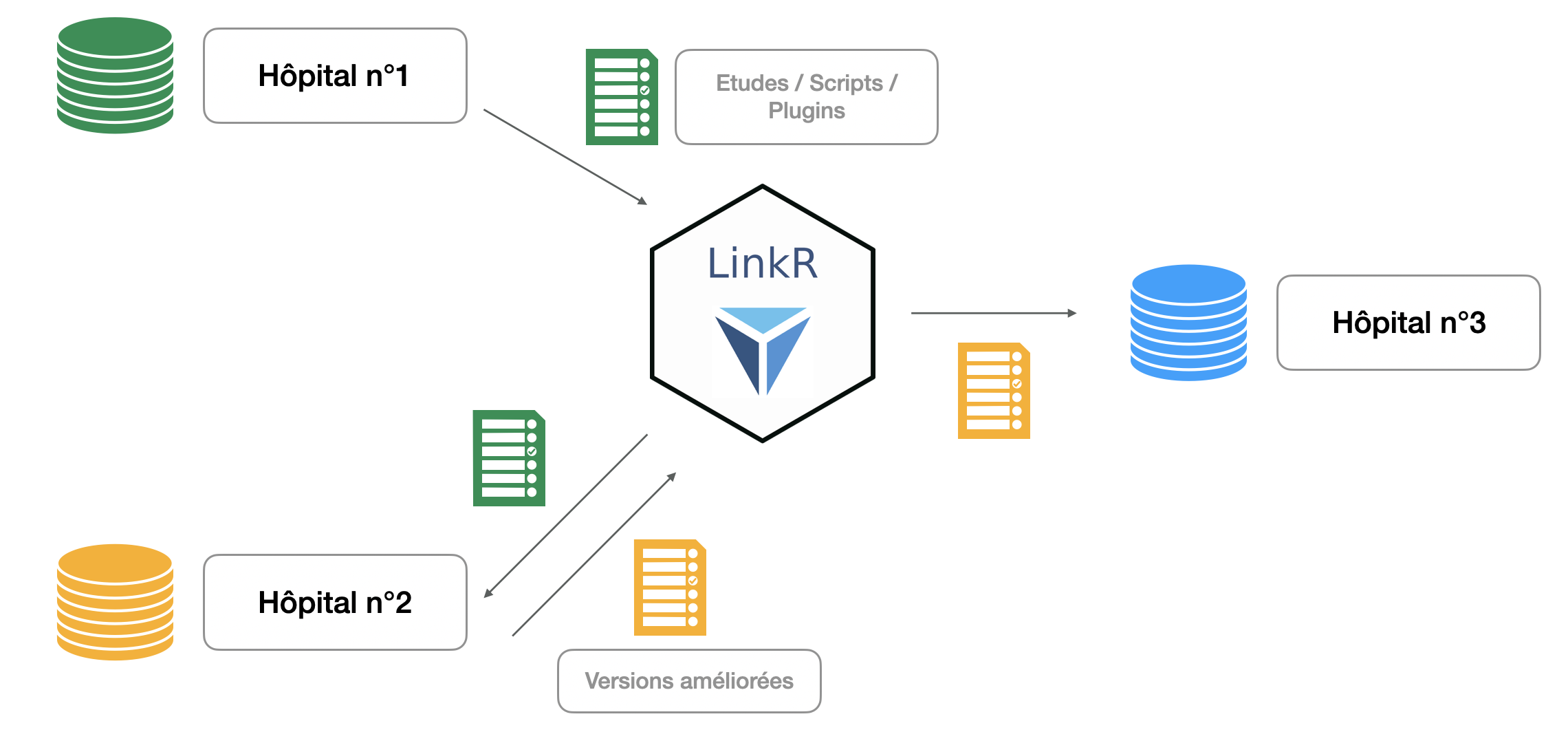
Interopérabilité algorithmique proposée par LinkR
En combinant l’interopérabilité des données ainsi que l’interopérabilité algorithmique (partage des études, scripts et plugins), LinkR permet rééllement de faire de la recherche selon les principes de la “Science Ouverte” ou open science2.
Ceci permet également une protection des données de santé : LinkR suit une architecture décentralisée, ce sont les algorithmes qui voyagent et non les données.

Catalogue de plugins disponibles sur le git d'InterHop. L'installation d'un plugin se fait en un clic.
LinkR est une application web créée avec la librairie Shiny en R.
C’est un logiciel open source, sous licence GPLv3.
L’application peut être installée via ShinyProxy et être protégée par une authentification par SSO.
Pour installer l’application, suivez les étapes indiquées ici.
Vous pouvez également tester l’application ici :
InterHop propose pour ses outils numériques un l’hébergement de données sur ses serveurs HDS (hébergeur de données de santé), loués chez GPLExpert.
L’association propose également d’utiliser Goupile, un outil d’eCRF, permettant de recueillir des données directement sur les serveurs HDS.
Toujours dans le cadre d’InterHop il est possible d’utiliser LinkR sur les serveurs HDS.
Ainsi, il sera possible de recueillir les données puis de réaliser les études au même endroit, de façon sécurisée.
InterHop propose aussi un support à l’utilisation de LinkR.
Le projet étant open source et réalisé par des bénévoles, toute contribution est la bienvenue !